Hoy os voy a hablar del uso del lenguaje R para el análisis de microarrays. Si no lo conocéis, R es un lenguaje de programación para análisis estadístico, que puede utilizarse a través de comandos o con alguna interfaz gráfica.
En la facultad lo utilizábamos en la asignatura de estadística, y la interfaz era muy parecida a la de Statgraphics, por lo que creí que era prácticamente lo mismo, para hacer estudios típicos de estadística. Pero en la asignatura que estoy estudiando ahora en el máster he visto que además de eso, es posible instalarle paquetes, en este caso especiales para el análisis de microarrays, pero supongo que para otras áreas también existirán paquetes especiales.
En mi caso he utilizado los paquetes del proyecto Bioconductor para detectar genes diferencialmente expresados, que se puede decir que son los que más se activan durante el estudio con el microarray (en un post anterior hablé de ellos). Además de éstos, por lo que he visto en la web, también tiene paquetes para realizar anotación y secuenciación de genomas.
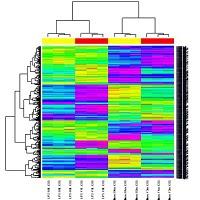
Los gráficos que se obtienen son de mucha calidad! El que he puesto en este post es uno de los del trabajo que hice para la asignatura, pero al guardarlo para web pierde mucho ;)
Realmente desconocía la potencia de este lenguaje, y las posibilidades que da en cuanto a adaptación a la búsqueda de solución de diferentes tipos de problemas.
Por lo que he podido ver, Matlab también dispone de herramientas para bioinformática, pero éste es de pago.
Y vosotros, ¿conocéis algún programa o lenguaje estadístico que también permita realizar análisis estadísticos más específicos?
Feliz fin de semana!!

