Uno de los problemas más graves a los que se puede enfrentar una célula, ya sea procariota o eucariota, es el daño a su material genético o DNA, tanto causado por factores externos (agentes químicos tóxicos, rayos UVA, radiactividad…) como por factores internos (actividad metabólica, agentes oxidativos…). De las muchas agresiones que sufre el DNA, que pueden llegar hasta un millón por célula y día, la más peligrosa es la rotura simultánea de las dos hebras de la doble hélice de DNA, ya que es el único caso en que un solo evento de daño, de no ser reparado, puede provocar fragmentación de los cromosomas, inestabilidad del genoma e incluso la muerte de la célula.
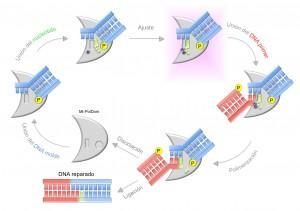
Mecanismo NHEJ
Las células poseen varios mecanismos de reparación del DNA, siendo dos los específicos para solucionar las roturas de doble cadena: recombinación homóloga y reunión de extremos no homólogos (non-homologous end-joining; NHEJ). La recombinación homóloga es el proceso de reparación con menor tasa de error para este tipo de daño, pero sólo puede ocurrir entre dos cromosomas hermanos, es decir, una vez que el material genético se ha duplicado durante un ciclo de división celular. Sin embargo, la mayoría de las células del organismo, que no se están dividiendo, no llegan a tener nunca dos copias disponibles de cada cromosoma, por lo que el mecanismo mayoritario para la reparación de roturas de doble cadena en células eucariotas es el de NHEJ, que realiza un empalme directo de los dos extremos de la rotura, normalmente a costa de generar una cierta tasa de mutación, pérdida o ganancia de información genética, especialmente cuando entre las secuencias de los dos extremos de DNA no existe ninguna complementariedad. Este mecanismo de reparación se encuentra conservado evolutivamente desde las bacterias hasta los mamíferos. En el laboratorio del Profesor Luis Blanco, en el CBMSO, se descubrieron en el año 2000 dos de las proteínas humanas implicadas en el mecanismo de NHEJ: las DNA polimerasas de la familia X Polλ y Polμ. Recientemente, algunos miembros del laboratorio se han embarcado en la tarea de estudiar la bioquímica de sus homólogos en Mycobacterium tuberculosis (Mt-PolDom), junto con el laboratorio de cristalografía del Profesor Aidan Doherty, en la Universidad de Sussex, y a consecuencia de esta colaboración se publicó un artículo en Science describiendo la sinapsis improductiva de dos extremos de DNA mediada por dos unidades de Mt-PolDom.
Los jugadores que intervienen en el proceso de NHEJ bacteriano son las proteínas Ku, que mantienen cerca los dos extremos de la rotura, y la Ligasa D, una proteína con aspiraciones de navaja suiza, ya que contiene tres dominios que facilitan todas las herramientas necesarias para recortar (nucleasa), rellenar (polimerasa) y pegar (ligasa) los extremos de la rotura. El dominio polimerasa (Mt-PolDom) es muy especial, ya que es el encargado de leer la secuencia genética de los extremos y decidir cómo unirlos, añadiendo como ladrillos los ribonucleótidos (rNTPs: A, C, G o U) necesarios para que no queden huecos en el DNA y se pierda la menor cantidad de información posible. Los rNTPs son mucho más abundantes que los nucleótidos y, por tanto, más accesibles para poder realizar una reparación rápida del DNA.
Las DNA polimerasas típicas, encargadas de generar copias del DNA, van creando las nuevas cadenas siempre en el mismo sentido, denominado 5’è3’. Pero no pueden iniciar la copia desde cero, sino que normalmente necesitan que otras enzimas, las primasas, sinteticen un pequeño trozo de RNA, llamado “primer”, que sirve para que la polimerasa se coloque y empiece a trabajar. Estas primasas inician la síntesis de este primer en sentido contrario al normal. Desde el punto de vista de su origen evolutivo, el PolDom de Mycobacterium tuberculosis pertenece a la familia de las primasas eucariotas, y en el artículo recientemente publicado en Molecular Cell por el Profesor Luis Blanco y sus colegas María José Martín y Raquel Juárez, en colaboración con el grupo inglés, se muestra la primera prueba de que Mt-PolDom ha conservado esta capacidad de generar trozos muy pequeños de material genético de tipo RNA, en el sentido contrario al normal. Esta característica única le confiere ventaja a la hora de usar sustratos tan especiales como lo son los de NHEJ, en los cuales la pieza que indica cual de los cuatro posibles ladrillos se introducirá (la base molde), está en un extremo de DNA distinto a aquel sobre el que debe colocarse el ladrillo (el extremo del primer). Siempre se había considerado que lo primero que debe colocarse en posición correcta sobre la polimerasa son ambos extremos de DNA, como si se tratase de dos folios ligeramente superpuestos, y una vez que éstos estuvieran alineados, ésta seleccionaría el nucleótido entrante, que actuaría como grapa. Sin embargo, en el nuevo complejo que se describe en el artículo, se observa que Mt-PolDom se encuentra cargada con la grapa y solamente uno de los folios, el que hace de molde director, mientras que el otro folio, aquel sobre el que tiene que colocarse la grapa (primer) no se encuentra en escena. En la estructura cristalográfica estudiada se muestra por primera vez un complejo enzimático de este tipo, denominado pre-ternario. Así, este trabajo pone sobre la mesa un nuevo fotograma que reordena completamente la película del proceso de NHEJ en bacterias, ayudándonos a entender un poco más el complejo mecanismo por el cual estos microorganismos patógenos se defienden de las agresiones externas.
Fdo.: Maria Jose Martín Pereira (CBMSO)
Referencia bibliográfica:
Nigel C. Brissett, Maria Jose Martin, Robert S. Pitcher, Julie Bianchi, Raquel Juarez, Andrew J. Green, Gavin C. Fox, Luis Blanco, Aidan J. Doherty. Structure of a novel pre-ternary complex involving a prokaryotic NHEJ polymerase. Molecular Cell (2011), doi:10.1016/j.molcel.2010.12.026.
