Os invito a ir poniendo en los comentarios datos sobre el coronavirus, en modo tormenta de ideas. Datos o ideas que creáis que pueden ser de utilidad en modo resumido, cortas, sin extenderse. Podéis acompañarlos de imágenes, y si van con referencia bibliográfica mejor que mejor (incluso poner solo algún paper que os haya resultado interesante).
Nada de reflexiones o desarrollos sobre teorías especulativas. Nada de política. Solo datos del reservorio del virus, mecanismo de infección, estructura, evolución desde otros tipos de virus,… datos científicos contrastados.
Muchas veces surgen ideas de este tipo de colaboraciones, formas de ver un problema desde otro punto de vista,.. otras veces no sucede nada.
Empiezo yo:
– en murciélagos hay una adaptación evolutiva del enzima STING para generar menos respuesta antiinflamatoria contra el virus y permitir una convivencia asintomática.
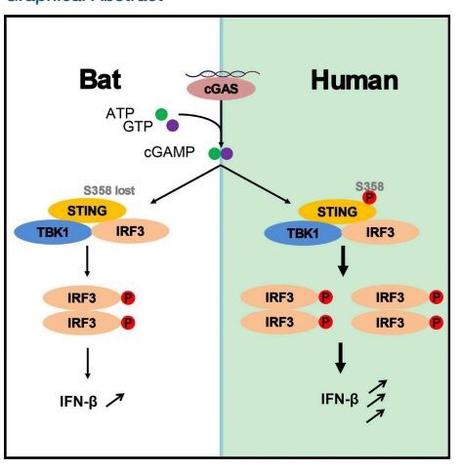
– la similitud con el virus SARS-Cov es de un 80%
– la polimerasa del coronavirus es muy similar a la del virus de la hepatitis.
– el receptor del SARS-Cov-2 es ECA2 (Enzima Convertidora de Angiotensina 2) el mismo receptor que usó el SARS-CoV en 2002.
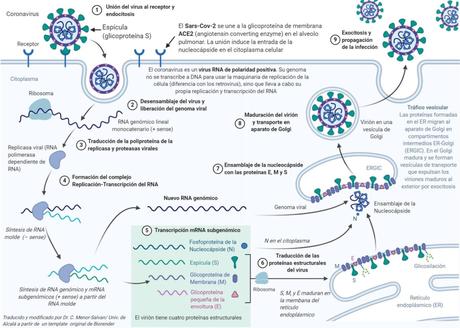
–
– En el anterior brote de SARS-CoV los animales de los “mercados húmedos” chinos que estaban infectados fueron las civetas, hurones y mapaches.
– La Enzima Convertidora de Angiotensina (ECA) convierte la angiotensina I en angiotensina II, ésta última se une al receptor AT1aR provocando daños en el tejido pulmonar y edema. La ECA2 (lugar donde se une el SARS-CoV-2) convierte la dañina angiotensina II en una forma menos lesiva llamada angiotensina 1-7. El SARS-CoV-2 se une a la ECA2 produciendo una disminución de esa enzima en las células afectadas. Los niveles de ECA no se ven afectados, la angiotensina II se incrementa y con ella el daño pulmonar.
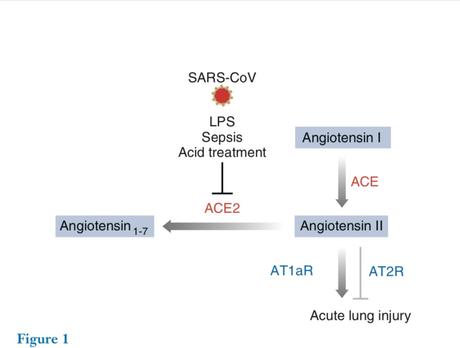
Cualquier aporte es bienvenido. A medida que vaya encontrando algún dato que me llame la atención iré actualizando el post (hay unos cuantos más en el borrador, pero con que pongamos 4-5 por tanda sobra).
Gracias por participar.
Fuentes:
– Xie, J., Li, Y., Shen, X., Goh, G., Zhu, Y., Cui, J., … Zhou, P. (2018). Dampened STING-Dependent Interferon Activation in Bats. Cell Host & Microbe, 23(3), 297–301.e4. doi:10.1016/j.chom.2018.01.006
– De Wit, E., van Doremalen, N., Falzarano, D., & Munster, V. J. (2016). SARS and MERS: recent insights into emerging coronaviruses. Nature Reviews Microbiology, 14(8), 523–534. doi:10.1038/nrmicro.2016.81
– Weston, S., & Frieman, M. B. (2020). COVID-19: Knowns, Unknowns, and Questions. mSphere, 5(2). doi:10.1128/mSphere.00203-20
– Chen, J., & Subbarao, K. (2007). The Immunobiology of SARS. Annual Review of Immunology, 25(1), 443–472. doi:10.1146/annurev.immunol.25.022106.141706
ó
