El cerebro es un órgano extremadamente complejo formado por diversos tipos de células entre las que destacan sus cerca de cien mil millones de neuronas. Éstas se interconectan formando una extensa y coordinada red tridimensional. Conocer cómo se desarrolla y cómo trabaja esta red es clave para estudiar el funcionamiento del cerebro y para entender enfermedades tan graves como el Alzheimer.
Estudiar el comportamiento de las redes neuronales se ha convertido en un verdadero reto para los investigadores. Las neuronas, lejos de trabajar de forma independiente, lo hacen en conjunto y, para comprender su funcionamiento, debemos observar regiones del cerebro completas manteniendo las redes neuronales intactas (1). Esto implica que para observar estas redes en el cerebro de un ratón deberíamos utilizar abordajes basados en la imagen médica como la Resonancia Magnética Nuclear (2) o la Tomografía por Emisión de Positrones (3). Estas técnicas poseen lo que denominamos una gran profundidad de imagen y con ello permiten mostrar un cerebro íntegro de manera tridimensional sin cirugía ni procesamiento alguno. Sin embargo, estas técnicas carecen de resolución de imagen, esto es, no consiguen observar objetos de muy reducido tamaño como son las neuronas.

Por otro lado, las técnicas de microscopía modernas y, más allá, la microscopía electrónica (4), son poseedoras de una enorme resolución y, por tanto, nos permitirían observar las diminutas neuronas con asombroso detalle.
Pero al mismo tiempo y en contrapartida, estas técnicas tienen muy limitada su profundidad de imagen y son incapaces de observar muestras gruesas y de profundizar en ellas para realizar reconstrucciones completas del tejido (5). Ni siquiera técnicas de microscopía avanzadas que mejoran la profundidad de imagen como la microscopía confocal pueden observar una muestra biológica más allá del medio milímetro de grosor. Con estas propiedades, la microscopía muestra el tejido a nivel celular pero únicamente en finas secciones procesadas las cuales están lejos de contener redes neuronales íntegras.
Así, las técnicas capaces de profundizar hasta 10 cm en un tejido no podrán mostrar objetos mucho menores de 0.1 mm y al mismo tiempo, las técnicas capaces de ver objetos de menos de un micrómetro serán incapaces de penetrar en el tejido más allá de medio milímetro.
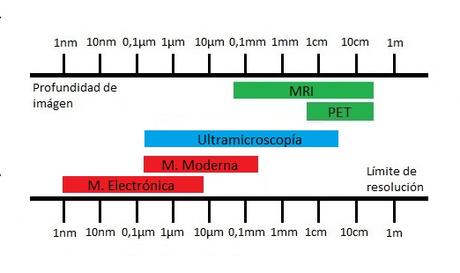
Profundidad de imagen y límite de resolución de técnicas de microscopia moderna y electrónica y de técnicas de imagen médica (Resonancia magnética Nuclear o MRI y Tomografía por emisión de Positrones o PET). Entre ellas, a modo de puente salvando el abismo, se encuentra la Ultramicroscopia.
Nos encontramos, parece, con un abismo insalvable: a un lado, técnicas que permiten obtener imágenes tridimensionales de órganos enteros incapaces de estudiar la composición celular. Al otro lado, técnicas capaces de analizar tejidos a nivel celular que no pueden observar una muestra completa de manera tridimensional.
El puente que salvaría esa caída es fácil de imaginar pero complejo de llevar a cabo: una técnica capaz de observar los componentes celulares de una muestra gruesa de tejido, es decir, capaz de realizar una imagen tridimensional de un cerebro de ratón entero en la cual se pudiesen diferenciar las redes neuronales a estudiar (6).
En el año 1902, Henry Siedentopf trabajaba como físico óptico para la compañía de imagen microscópica Carl Zeiss y Richard Zsigmondy era profesor de química inorgánica de la Universidad de Göttingen. Ambos estudiaban unas diminutas partículas denominadas Ultramicronen y, para poder observarlas de manera fiable, necesitaron idear una técnica de microscopía a la que llamaron ultramicroscopia (7).
En la microscopía tradicional, la muestra se observa y se ilumina en el eje vertical, es decir, se observa e ilumina desde arriba, sin embargo, en la ultramicroscopía la iluminación y la observación de la muestra están en diferentes ejes y, mientras que se observa normalmente desde arriba, la muestra es iluminada desde los laterales (8). Además, Siedentopf y Zsigmondy idearon un conjunto de lentes que permitía generar un finísimo plano de luz que iluminaba solo una lámina de la muestra. Su ingenio les llevó a elaborar el primer ultramicroscopio el cual le valió a Zsigmondy el Premio Nobel de Química en 1925 (7).
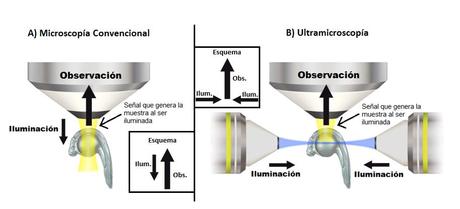
Comparación entre las bases de la microscopía convencional (A) y la ultramicroscopía (B). Mientras que en la microscopía convencional se ilumina y observa la muestra en el mismo eje vertical (esquema), en la ultramicroscopía se genera un fino haz lumínico que ilumina la muestra desde los laterales mientras la señal generada se observa en un ángulo de 90º desde arriba. Adaptada de MicroscopyU: The Source of Microscopy Education by Nikon. Nikon.
El ultramicroscopio fue inicialmente ideado para observar las diminutas Ultramicronen y no para observar tejido biológico alguno. Por esto, su uso permaneció de manera exclusiva dentro del mundo de la química inorgánica y nadie, durante décadas, se atrevió a observar muestras biológicas utilizando sus bases. Además, para ser utilizada en biología, esta técnica requiere de la transparencia de las muestras (5) y aunque el Dr. Spalteholz, anatomista alemán, ya ideó protocolos de clarificado de tejido biológico a inicios del siglo XX (5), no fue hasta finales del mismo siglo cuando las dos técnicas convergieron.
Concretamente, fue el Dr. Arne H. Voie y sus colaboradores, en 1993, los primeros en introducir la creación de Siedentopf y Zsigmondy en la observación de muestras biológicas transparentadas.
Voie pretendía reproducir con gran resolución la estructura de la cóclea, parte indispensable del aparato auditivo, con intención de mejorar los implantes para tratar pérdidas auditivas. Para esto, partió de las técnicas de transparentado tisular y de las bases de la ultramicroscopía (ambas de inicios de siglo) y, beneficiándose de la óptica y la química modernas así como del láser, transparentó una cóclea y la iluminó de forma perpendicular con un finísimo plano de luz. Acto seguido movió la muestra a lo largo del láser, como si de un escaneo se tratase, consiguiendo como resultado una reproducción tridimensional de la cóclea en su conjunto.
El Dr. Voie acababa de sentar los cimientos para construir el puente que salvaría un importante abismo técnico dentro de la investigación biológica. La unificación de la ultramicroscopía de Siedentopf-Zsigmondy y la transparentación de Spalteholz daría a luz a la ultramicroscopía moderna la cual entraría el campo de la biomedicina y pasaría a conocerse internacionalmente como Ligh-Sheet Microscopy o ultramicroscopía.
Esos cimientos colocados por Voie tardarían en ampliarse y no fue hasta 2004 cuando empezó la verdadera construcción del puente sobre el abismo. En ese año, el Dr. Huisken, del EMBL en Heidelberg, publicó una mejora de la técnica denominada SPIM, con la cual observó dos modelos de investigación clave para la biomedicina: un embrión de pez Medaka y el desarrollo embrionario de la mosca Drosophila.
No te pierdas el siguiente video de un embrión de pez Medaka (Huisken et al., 2004).
http://www.acercaciencia.com/wp-content/uploads/2017/11/Video.1_Pez_Medaka.mp4Durante los 5 años siguientes se vivió una verdadera revolución, dominada principalmente por los investigadores Becker, Dodt y Jährling, de la Universidad Técnica de Viena. Durante estos años se pusieron a punto protocolos que, de manera tridimensional y con importante resolución, permitieron observar moscas Drosophila adultas e incluso embriones completos de ratón.
En el siguiente video se puede observar una reconstrucción en 3D de un embrión de ratón (Adaptado de Becker et al., 2008).
http://www.acercaciencia.com/wp-content/uploads/2017/11/Video.4_Embrión_Entero.mp4Tras esto, pronto se consiguieron observar redes de vasos sanguíneos en órganos como el corazón o el cerebro, pudiendo analizar desde vasos grandes y arteriolas hasta la red microvascular y capilar de los tejidos. Y al mismo tiempo que fuimos capaces de observar las redes vasculares, comenzamos a alcanzar el tan deseado hito para los investigadores en neurociencia: la observación de redes neuronales. De hecho, uno de los pioneros fue el Dr. Dodt, mostrando regiones enteras del hipocampo de un cerebro a la vez que era capaz de detectar individualmente las neuronas que lo componían.
En el siguiente video se pueden ver regiones enteras de hipocampo de ratón (Dodt et al., 2007)
http://www.acercaciencia.com/wp-content/uploads/2017/11/Video.6_Cerebro_Dodt.mp4Durante la última década, la ultramicroscopía o Light-Sheet Microscopy ha ido adquiriendo mejoras dentro del propio microscopio (óptica, láseres, objetivos) al mismo tiempo que mejoraba imparable la transparentación tisular, parte crucial de la eficiencia de esta técnica. Además, se ha mejorado también el complejísimo procesamiento informático de las intrincadas imágenes tridimensionales que se obtienen con este tipo de microscopía.
De la mano de nuevos investigadores como la Dra. Viviana Gradinaru, del Instituto de Tecnología de California, la ultramicroscopía ha generado durante los últimos años impactantes visualizaciones tridimensionales del cerebro en su conjunto con una resolución sin precedentes (Vídeo.7). La propia Dra. Gradinaru, junto con Karl Deisseroth, han conseguido incluso trazar de manera individual una neurona a través de una red en un cerebro humano. Todos estos logros durante los últimos años le han valido a la ultramicroscopía una atención central en muchos campos dentro de la comunidad científica y la prestigiosa revista científica Nature consideró este método como el método del año en 2014 (9). En cuanto al futuro, mejor tener los ojos bien abiertos para no perdernos los seguro impresionantes mundos que la ultramicroscopía será capaz de mostrarnos.
Bibliografía consultada
1. Keller PJ, Ahrens MB, Freeman J. Light-sheet imaging for systems neuroscience. Nat Methods. 2015 Jan;12(1):27-9.
2. El Tamiz: ¿En qué consiste una Resonancia Magnética Nuclear (RMN)?
3. El Tamiz: ¿En qué consiste una Tomografía por Emisión de Positrones (TEP)?
4. Hipertextos del Área de Biología: Microscopía electrónica: Barrido y Transmisión
5. AcercaCiencia: La fisicoquímica de la trasparencia.
6. Becker K, Jährling N, Saghafi S, Dodt HU. Ultramicroscopy: light-sheet-based microscopy for imaging centimeter-sized objects with micrometer resolution. Cold Spring Harb Protoc. 2013 Aug 1;2013(8):704-13.
7. Söderbaum, H.G. Award ceremony speech (1926). Nobelprize.org
8. MicroscopyU – The source for microscopy education – Nikon: Light Sheet Fluorescence Microscopy.
9. Nature Methods: Method of the Year 2014.
Referencias videos
-Video embrión pez Medaka: Huisken J, Swoger J, Del Bene F, Wittbrodt J, Stelzer EH. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 2004 Aug 13;305(5686):1007-9.
-Video 3D embrión ratón: Adaptado de imágenes de: Becker K, Jährling N, Kramer ER, Schnorrer F, Dodt HU. Ultramicroscopy: 3D reconstruction of large microscopical specimens. J Biophotonics. 2008 Mar;1(1):36-42.
-Video hipocampo ratón: Dodt HU, Leischner U, Schierloh A, Jährling N, Mauch CP, Deininger K, Deussing JM, Eder M, Zieglgänsberger W, Becker K. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nat Methods. 2007 Apr;4(4):331-6.

Graduado en Biquímica y Biología Molecular.
“He visto como la ciencia se muere si la dejas mucho tiempo en un laboratorio, por eso estoy comprometido a sacarla de vez en cuando a la calle”.
Álvaro Sahún Español
Si te ha gustado el artículo te pedimos que nos ayudes a difundirlo. Compartir el conocimiento hace al cambio. Puedes hacerlo mediante la botonera que está debajo. ¡Gracias!

